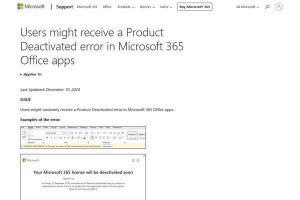
京都大学(京大)は5月18日、DNA一本鎖切断酵素であるCas9 Nickaseを用いたゲノム編集法 CRISPR/Nickaseシステムを確立したと発表した。
同成果は、京都大学大学院農学研究科 植田充美教授、黒田浩一准教授、博士課程の里村淳氏らの研究グループによるもので、5月18日付けの英国科学誌「Scientific Reports」に掲載された。
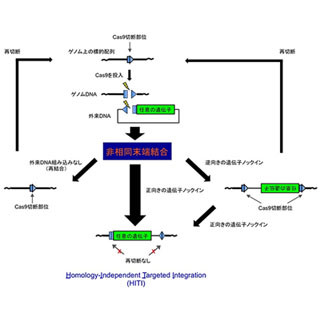
近年、ゲノム編集法としてCRISPR/Cas9システムが注目を集めている。同システムでは、ガイドRNAがPAM配列(NGG:Nは任意の塩基)に続く20塩基を認識して、Cas9がDNAを切断する。この切断領域に相補的なドナーDNAを加えると相同組換えにより任意の配列をノックインでき、非相同末端結合により切断配列をランダムに修復するとノックアウトが可能になる。
しかし、同システムが正確に編集できる領域は、Cas9が認識できるNGG配列とそれに続く20塩基のみであり、これがゲノム中に存在するのは60%程度である。また、ガイドRNA認識配列外の塩基を編集する場合、相同組換え後もガイドRNA認識配列が残るため、Cas9ヌクレアーゼにより、ゲノムが繰り返し切断・修復された結果、最終的には非相同末端結合により望まない塩基の挿入、欠失が生じてしまうという課題もある。
そこで同研究グループは今回、一本鎖切断酵素であるCas9 Nickaseに着目。ゲノムワイドで高効率な編集技術CRISPR/Nickaseシステムを構築した。Cas9 Nickaseで生じる一本鎖切断(ニック)は、正確に修復されるうえ、旧来のCas9により生じる二本鎖切断と異なり、相同組換えを誘導するが非相同末端結合は誘導しない。そのため、Nickaseによる相同組換え後に標的配列が残ったときでも、ニックが再度導入されるだけで、非相同末端結合による余分な配列の挿入や欠損は起こらないことになる。
また、CRISPR/Nickaseシステムは、ニックから50塩基対離れた領域でも正確に編集することができ、理論上は、CRISPR/Cas9よりも40%以上も広いゲノム領域、すなわち、ほぼゲノム全域を編集できることを示している。
同研究グループは、今回の成果について、ゲノム上のほぼすべてのSNPs(一塩基多型)の修復編集が可能になるとともに、目的どおりのSNPsの導入も可能になることから、これからのゲノム科学の発展に貢献できると説明している。