理化学研究所(理研)は4月12日、日本人300例の肝臓がんの全ゲノムシーケンス解析を実施し、それらのゲノム情報をすべて解読したと発表した。
同成果は、理研 統合生命医科学研究センター ゲノムシーケンス解析研究チーム 中川英刀チームリーダー、藤本明洋副チームリーダー、国立がん研究センター がんゲノミクス研究分野 柴田龍弘分野長、十時泰ユニット長、東京大学 医科学研究所附属 ヒトゲノム解析センター 宮野悟教授、広島大学大学院 医歯薬保健学研究院 茶山一彰教授らの研究グループによるもので、4月11日付けの米科学誌「Nature Genetics」に掲載された。
肝臓がんの主な原因は、肝炎ウイルスの持続感染であり、B型(HBV)やC型肝炎ウイルス(HCV)の感染に伴う慢性肝炎から、肝硬変を経て発症する。肝臓がんの治療法にはさまざまな方法があるが、その効果は十分ではなく、ゲノム情報に基づく発がん分子メカニズムの解明と新たな治療法や予防法の開発が求められている。
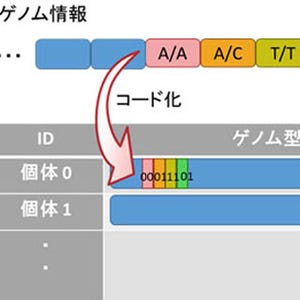
今回、同研究グループは、主にウイルス肝炎を背景として発症した日本人300例の肝臓がん組織と同一患者由来の血液からDNAとRNAを抽出し、それらの全ゲノム情報を最新の次世代シーケンサーと東京大学ヒトゲノム解析センターの生命科学専用スーパーコンピュータ「SHIROKANE」を利用して解読。肝臓がんに起きているゲノム変異をすべて同定した。解析した塩基配列数は全部で約70兆塩基、情報量としては約300TB以上となる。
この結果、ひとつの腫瘍あたりのゲノム異常は平均で約1万カ所あり、塩基がひとつだけ変わる「点突然変異」以外に、さまざまな形態の変異を検出。特に、既知のがん関連遺伝子(p16、APC、TERT、CCND1、RB1など)に加えて、新規のがん関連遺伝子(ASH1L、NCOR1、MACROD2、TTC28など)の染色体構造異常や点突然変異を多数発見した。
また、HBVとアデノ随伴ウイルスゲノムの肝臓がんゲノムへの組み込みも検出。これらのウイルスゲノム挿入によって組み込み部位周辺の遺伝子の発現が変化していることが明らかになった。さらに、遺伝子発現や構造に影響を及ぼす可能性のある非コード領域の変異や非コードRNAの変異も多数検出。特に、染色体の先端構造を維持して細胞の不死化や染色体の安定性に関わる酵素をコードしている「TERT遺伝子」においては、ゲノム構造異常、ウイルスゲノムの組み込み、プロモーター領域の点突然変異、コピー数異常といったさまざまなタイプのゲノム変異が集積しており、肝臓がんにおけるTERT遺伝子の発現を恒常的に上昇させていた。
これら多数のゲノム変異情報を用いると、肝臓がんは大きく6つに分類され、がん抑制遺伝子「TP53」に関わる遺伝子群に変異がある肝臓がんは予後が不良であるなど、肝臓がん予後の生存率がこれらの分類によって大きく異なっていることも明らかになった。
同研究グループは、今後、同成果ががんのゲノム配列情報に基づいた肝臓がん治療の個別化や新規の治療法・予防法開発へ発展する可能性があるとしている。