理化学研究所(理研)は7月25日、細胞1個が発現する遺伝子を網羅的に定量化する方法「Quartz-Seq(クオーツセック)法」を開発し、同じ種類の細胞で、かつ同じ「細胞周期」にある細胞同士でも、細胞ごとに遺伝子発現のパターンに差があることを明らかにしたと発表した。
成果は、理研 生命システム研究センター 合成生物学研究チームの上田泰己グループディレクターらの研究チームによるもの。研究の詳細な内容は、4月17日付けで英科学誌「Genome Biology」に掲載済みだ。
遺伝子とは、主に生物の体を構成するタンパク質に変換される遺伝情報を持ったDNA配列のことだ。そのDNA配列が転写と呼ばれる過程を経てメッセンジャーRNA(mRNA)に変換され、続いてmRNAの情報を基にタンパク質が合成されるのである。この一連の過程の中でmRNAは遺伝子発現の有無を確認できる産物として重要な存在だが、細胞が含んでいるmRNAの総量はごく微量で、10pg程度しかなく、1つひとつの細胞について、その遺伝子発現を精度よく定量化することは困難だった。
遺伝子発現パターンを網羅的に定量化する方法として、現在用いられているのが「1細胞RNAシーケンス法」だ。この手法は、細胞ごとの遺伝子発現の不均一性を計測するための有効な手段になり得るが、現状では感度や再現性が十分ではなく、実験の簡便さにも欠けるという問題があった。具体的には、遺伝子から転写されたmRNAに対応するcDNA(相補的DNA)の増幅が必要であり、理想的にはタンパク質に変換されるすべてのmRNAをcDNAに変換し、偏りなく増幅することが求められるのである。そのため、細胞1個の遺伝子発現パターンを定量化するための高精度で操作しやすい方法の開発が望まれていた。
そこで研究チームは、まず1細胞RNAシーケンス法に関して、「逆転写反応」、「PCR反応」、「逆転写プライマー由来副産物の軽減」という以下の3点について最適な条件を検討した(画像1~3)。
1. 逆転写反応
cDNAは、逆転写反応と呼ばれるmRNAをDNAに変換する反応で合成できる。具体的には、遺伝子から転写されたRNA配列の特定の位置に「プライマー」が結合し、そのプライマーを目印にDNA合成酵素が結合し、cDNAが作られる仕組みだ。研究チームは、プライマーをRNAに結合させるときの温度である「アニーリング温度」を最適化することで、全体的に偏りなくcDNAを増幅させることに成功した。
2.PCR反応
増幅能力が強いDNA合成酵素をcDNAの増幅反応に用いることで、発現頻度の低いmRNAも、発現頻度の多いmRNAと同様の効率で増幅させることに成功。
3.逆転写プライマー由来副産物の軽減
反応液中に残っているプライマー自体の増幅を防ぐため、逆転写反応後に、反応に使われなかった不要なプライマーを特定の酵素によってある程度分解し、除去する。さらに残ったプライマーの末端同士が結合しやすいような(「自己アニーリング」)配列を設計した。自己アニーリングした残存物の増幅は起こらないので、効果的に副産物の生成を軽減することに成功した。
なおプライマーとは、DNAを増幅するPCR法において用いられる化学合成した短い(20塩基程度)の「オリゴヌクレオチド(核酸)」である。鋳型となるDNAに相補的に結合したプライマーに新たなヌクレオチドが付加されることでcDNAが合成される。
これら3つのポイントを最適化することで、1細胞RNAシーケンス法をより高精度に改良した「Quartz-Seq(クオーツセック)法」が確立されたのである。Quartz-Seq法は従来法の「Smart-Seq(スマートセック)法」が必要とするゲル精製などの作業プロセスがなく、すべての反応を1つのマイクロチューブの中で行えるので操作が簡便になった点が大きな特徴だ。
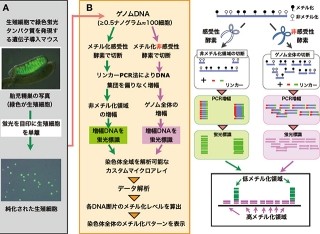
画像4は、Quartz-Seq法の概略で、以下に記す通り大まかに6つの流れがある。
- オリゴdT配列を持つ逆転写プライマーを用いて逆転写を行いmRNAに相補的なcDNAを得る
- 過剰な逆転写プライマーは「エキソヌクレアーゼI」で除去するが、完全に除くことはできない
- 逆転写されたcDNAの3'末端に「ターミナルトランスフェラーゼ」と呼ばれる酵素を用いて「poly-A付加」を行う。除ききれなかった逆転写プライマーも同様に修飾される
- Poly-A鎖に結合させた「タギングプライマー」からの伸長反応により相補的な鎖(第2鎖)を合成する。結果、両端に相補的配列がついたDNAとなる
- 逆転写プライマー由来の短いDNAの場合、両端の相補配列により残存プライマーはフライパン構造をとることで、「PCRプライマー」の結合が阻害される。一方でRNA由来の長いcDNAの場合、両端の相補配列が結合する前にPCRプライマーが結合することができる。結果として、逆転写プライマー由来の副産物の増幅は抑制され、目的のRNA由来cDNAの増幅が効率よく進む。
- 増幅したcDNAを精製する
Quartz-Seq法とSmart-Seq法を比較するため、1個のマウスES細胞由来のRNA(10pg)を用いた検証が行われた。その結果、再現性の相関係数はQuartz-Seq法で0.93、Smart-Seq法で0.7となり、Quartz-Seq法はSmart-Seq法と比べて高精度化を達成したのである。また2回の独立した実験で、約8000の遺伝子をとらえることができ、再現性があることも確認された。
これは、1細胞ではなく大量の細胞に相当する1μgのmRNAを用いた通常法で検出される遺伝子の内、9割をとらえている計算になる。一方、Smart-Seq法では、同様の実験で通常法の4割ほどの遺伝子しか検出できなかった。Quartz-SeqはSmart-Seq法と比べて再現性、検出感度の両面で傑出しているというわけだ。
次に、Quartz-Seq法を用いて異なる条件における細胞間での遺伝子発現パターンの違いの検出が試みられた。
1. 細胞種の違い
マウスES細胞と、そこから分化した細胞である「PrE(原始内胚葉)細胞」の遺伝子発現パターンが計測されて解析された。その結果、2つの細胞種間の遺伝子発現パターンを明瞭に区別することに成功した。
2. 同じ培養環境にある細胞が持つ細胞周期の違い
同じマウスES細胞について、細胞周期の異なる細胞間で遺伝子発現パターンが解析された。その結果、細胞種の違いと比べると小さいながらも細胞周期による遺伝子発現パターンの違いを検出することに成功したのである。なお細胞周期とは、細胞分裂で母細胞から生じた娘細胞が、再び母細胞となって再び細胞分裂を行い、新しい娘細胞になるまでの過程をいう。
3. 同一の培養環境かつ同じ細胞周期の細胞間の違い
細胞周期がG1期にあるマウスES細胞同士で解析した結果、遺伝子発現パターンにゆらぎが検出された。いくつかの遺伝子の発現が「非増幅1細胞qPCR法」(PCR増幅産物の増加をリアルタイムで計測してcDNAの定量を行う技術)を用いて測定され、ゆらぎの度合いが、Quartz-Seq法で得られた結果と相関していることが確認された。すなわち、これまで定量化することが難しかった細胞間の遺伝子発現パターンのゆらぎを定量的にとらえることができたというわけである(画像5・6)。
以上の結果から、3つの異なるレベルの細胞の違いを検出することに成功した。
|
|
|
|
網羅的遺伝子発現のゆらぎの検出。画像5(左):Quartz-Seq法による網羅的遺伝子発現のゆらぎの再現性。約17,000遺伝子のゆらぎに有為な再現性があった。画像6:非増幅1細胞qPCR法とQuartz-Seq法の遺伝子発現のゆらぎの相関。非増幅1細胞qPCR法によってQuartz-Seq法がとらえているものが遺伝子発現のゆらぎであることがわかった。Rは相関係数 |
|
Quartz-Seq法は簡便で高精度なため、多くのユーザーが容易に導入可能である点も特徴の1つだ。それにより、さまざまな分野での応用が期待できるという。例えば、免疫反応は性質の異なる多数の細胞間の相互作用で引き起こされるが、Quartz-Seq法はこれらの細胞間の遺伝子発現パターンの違いを簡単にとらえることができるため、新しい分子マーカーの発見につながる可能性がある。
また、正常な発生過程における細胞分化は同一の細胞間に生じる遺伝子発現の微妙なゆらぎが引き金になっているとも考えられているという。Quartz-Seq法によって、細胞間の遺伝子発現のゆらぎを計測することで、発生・分化のメカニズムの解明や薬剤・環境応答メカニズムの解明に寄与することが考えられるとした。
さらに、再生医療におけるiPS細胞とその分化細胞の不均一性・均一性の検証による品質管理、遺伝子発現の不均一性を起因とするがん細胞の悪性化や薬剤耐性化のメカニズム解明などの医療分野に貢献すると期待できるとしている。