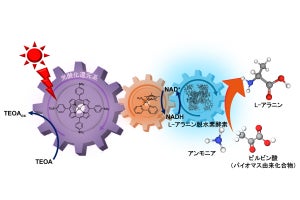
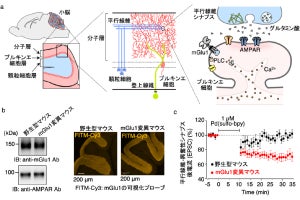
一方、SaCas9を標的DNA配列まで運ぶ役割を担うRNAと結合させた「SaCas9-RNA複合体」においては、2つの球状ドメインが互いに向かい合う構造を採ることがわかった。このRNAによるCas9の立体構造の安定化はSpCas9でも見られ、RNAがタンパク質の立体構造の安定化に重要な役割を担うことを強くサポートする結果となったとする。またSaCas9では、2つの向かい合った球状ドメインが閉じたり開いたりする動きを持つことも確認された。
-
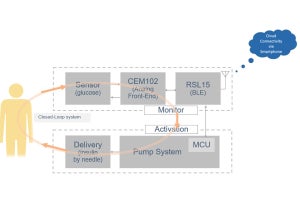
(上段)SaCas9単体の高速AFM動画。(中段)SaCas9-RNA複合体の高速AFM動画。(下段)SaCas9-RNA-DNA複合体のDNA切断時の高速AFM画像(出所:金大プレスリリースPDF)
さらに、標的DNAに結合した「SaCas9-RNA-DNA結合状態」では、三つ葉のクローバーのような構造が形成され、すべてのドメインにおいて安定な構造を形成することも明らかになった。
そして、SpCas9の場合では、DNA切断に重要な「ハサミ」部分であるHNHドメインがフラフラとよく動く様子が観察されていたが、SaCas9ではそのような様子は見られず、固く安定した立体構造を形成することがわかったという。さらにDNAの切断時には、HNHドメインが切断部位まで移動し、DNA切断後すぐにDNAから離れる様子が観察されたとする。
これまでの研究から、SaCas9は1分子において何度もDNAを切断できる機能を持つことが報告されており、高速AFMによって捉えられた今回の現象は、SaCas9の機能をサポートする結果であると考えられるとしている。それに加えて今回は、高速AFM基板上でSaCas9-RNA複合体が標的配列DNAへ結合する瞬間の画像も捉え、SaCas9における新たなDNA結合メカニズムの可能性も示されたという。
研究チームは、今回の研究によって、CRISPR-Cas9によるDNA切断ダイナミクスの統一的な理解を得ることができたとする。また、今回仮定された標的DNA探索の分子メカニズムを検証するために、今後、ほかの種に由来するCas9やその変異体を含めた、さらなる実験結果の積み上げが必要となるとしている。そして、今回得られた動的な構造情報が、より安全なゲノム編集ツールを開発するための基盤となることが期待されるとした。